20 相关性热图
20.1 什么是相关性热图?
相关性分析是指对两个或多个具备相关性的变量元素进行分析,从而衡量两个变量因素的相关密切程度。相关性的元素之间需要存在一定的联系或者概率才可以进行相关性分析。
当两个变量之间存在非常强烈的相互依赖关系的时候,我们就可以说两个变量之间存在高度相关性。若两组的值一起增大,我们称之为正相关,若一组的值增大时,另一组的值减小,我们称之为负相关。
默认一般使用皮尔逊算法算相关性。皮尔逊相关系数广泛用于度量两个变量之间的相关程度,其值介于-1与1之间。
计算完相关性后,我们通过热图做可视化。热图(heatmap)是一种将规则化矩阵数据转换成颜色色调的常用的可视化方法,其中每个单元格对应数据的某些属性,属性的值通过颜色映射转换为不同色调并按规则填充单元格。


本文我们就来讨论一下相关性热图是如何绘制的以及如何对其进行解读。
20.2 绘图前的数据准备
demo数据可以在https://www.bioladder.cn/shiny/zyp/bioladder2/demoData/Cor/demo.txt下载。
包含2个维度的数据,通常情况下每一行代表一个基因,每一列代表一个样本。
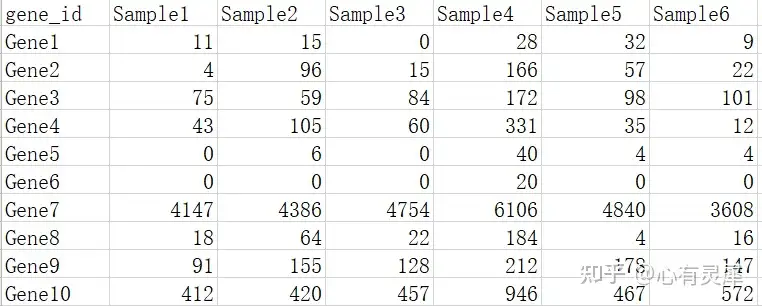
20.3 R语言怎么画相关性热图
# 加载R包,没有安装请先安装 install.packages("包名")
library(pheatmap)
# 读取相关性热图数据文件
df= read.delim("https://www.bioladder.cn/shiny/zyp/bioladder2/demoData/Cor/demo.txt",# 这里读取了网络上的demo数据,将此处换成你自己电脑里的文件
header = T,
row.names = 1
)
# 计算相关性
r <- cor(df,
method = "pearson", # 计算相关性的方法有"pearson", "spearman", "kendall"
use = "pairwise.complete.obs" # 缺失值处理的方式
)
# 绘制热图
pheatmap(r,
show_colnames = TRUE, # 是否显示列名
show_rownames=TRUE, # 是否显示行名
fontsize=5, # 字体大小
color = colorRampPalette(c('#0000ff','#ffffff','#ff0000'))(50), # 指定热图的颜色
annotation_legend=TRUE, # 是否显示图例
border_color=NA, # 边框颜色 NA表示没有
scale="none", # 指定归一化的方式。"row"按行归一化,"column"按列归一化,"none"不处理
cluster_rows = TRUE, # 是否对行聚类
cluster_cols = TRUE # 是否对列聚类
)
# 更多参数可以输入 ?pheatmap 查看20.4 相关性热图结果解读
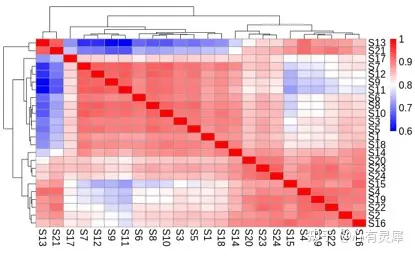
如图,是一副组学研究中热图的常用绘制模式,每个小方格表示每个样本之间的相关系数,其颜色表示相关性大小,相关性越大颜色越深。聚类树形图表示对来自不同实验分组的不同样品的聚类分析结果。如果不聚类,排序将保持文件数据的默认方式。
20.5 BioLadder生信云平台在线绘制相关性热图
不想写代码?可以用BioLadder生信云平台在线绘制相关性热图。
网址: