6 差异分析
6.1 什么是差异分析?
差异分析就是分析两组数据是否有差异。
1.什么是FC?
翻译成中文是差异倍数(fold change),也叫作radio,简单来说就是基因在一组样品中的表达值的均值除以其在另一组样品中的表达值的均值。如果规定FC>2为上调,那么FC<1/2为下调。
2.什么是Pvalue?
统计检验获得的是否统计差异显著的一个衡量值,约定成俗的P-value<0.05为统计检验显著的常规标准。
3.什么是FDR?
FDR (false discovery rate),即校正后的P值,中文一般译作错误发现率。
6.2 数据准备
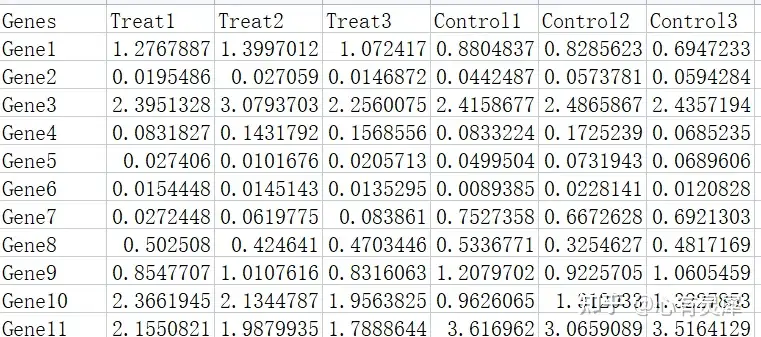
6.2.1 样本表达量信息
通常数据来源于搜库结果的定量表,每一行为一个基因,每一列是一个样本,数值为基因在对应样本中的表达量。
6.2.2 样本分组信息
包含2列数据,第一列为样本名称,第二列为分组名称。(本程序暂只支持2组)

6.3 R语言如何做差异分析(2组)
6.3.1 整理数据
library(tidyverse)
library(rstatix)
# 读取样本表达量信息
dfData = read.delim("https://www.bioladder.cn/shiny/zyp/bioladder2/demoData/variationAnalysis/data.txt")
# 读取样本分组信息
dfClass = read.delim("https://www.bioladder.cn/shiny/zyp/bioladder2/demoData/variationAnalysis/class.txt")
# 表达量+分组 合并,整理
df = dfData %>%
as_tibble() %>%
pivot_longer(-1,names_to = "Sample",values_to = "value") %>% # 转换成长数据
left_join(dfClass,by=c("Sample" = "Sample")) # 与分组数据合并
df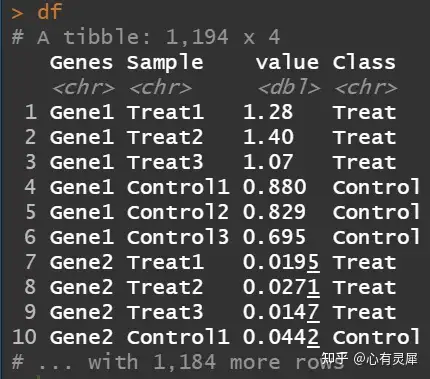
6.3.2 计算FC
dfFC = df %>%
group_by(Genes,Class) %>%
summarise(mean = mean(value,na.rm=T)) %>% # 计算平均值
pivot_wider(names_from = Class,values_from = mean) %>% # 转换成宽数据
summarise(FC = Treat/Control) # 实验组/对照组 计算差异倍数FC
dfFC
6.3.3 t_test 计算P 值
dfP = df %>%
group_by(Genes) %>%
t_test(value ~ Class,var.equal=T) # t_test 方法源于rstatix包; var.equal=T等方差
dfP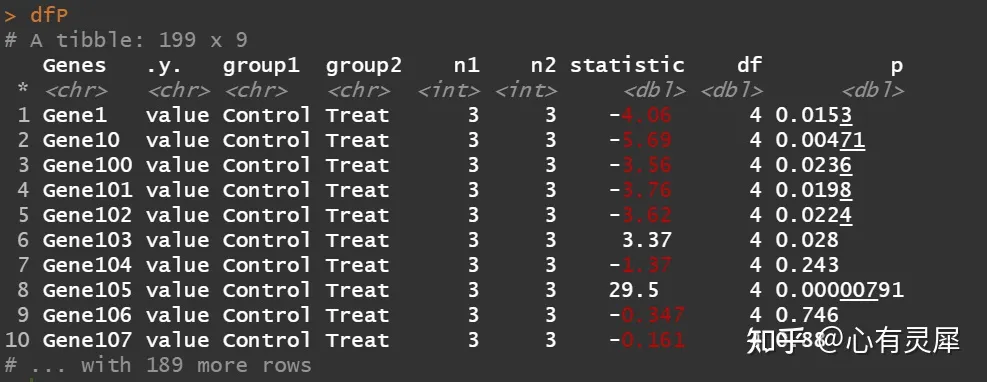
6.3.4 对P值FDR校正
dfP_FDR = dfP %>%
select(1,last_col()) %>%
mutate(FDR = p.adjust(.$p,method = "BH")) # p.adjust对P值FDR校正,算法选择BH
dfP_FDR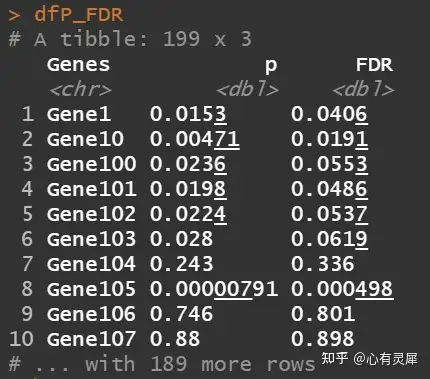
6.3.5 整理合并表格
dfData %>%
as_tibble() %>%
left_join(dfFC) %>%
left_join(dfP_FDR)
6.3.6 绘制火山图
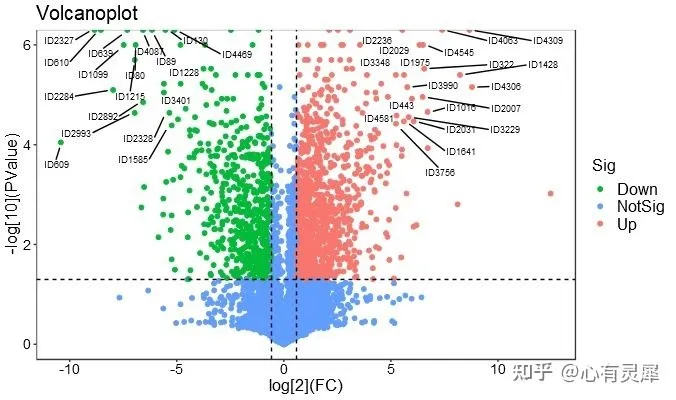
详细代码可参考另一篇文章,
6.4 BioLadder生信云平台在线做差异分析
不想写代码?可以用BioLadder生信云平台在线在线做差异分析。
免费使用,注册登录后畅享50+模块。
网址: